科研进展
《Genome Biology》发表多倍体团队“Small RNAs mediate transgenerational inheritance of genome-wide trans-acting epialleles in maize”
发布人: 发布日期: 2022-02-22 浏览次数:
杂交和回交是常用的动、植物育种手段。杂交引起的父母基因互作导致遗传和表观遗传变异,比如经典的副突变(paramutation)可以跨代遗传。但是,由表观遗传引起的跨代遗传的机理缺乏全基因组水平上的研究。2月9日,beat365平台多倍体杂种优势研究团队在Genome Biology(IF=13.583)上发表了题为Small RNAs mediate transgenerational inheritance of genome-wide trans-acting epialleles in maize的研究论文,揭示了玉米杂交诱导全基因组可跨代遗传的表观遗传现象及其调控机制。

表观遗传变异不受DNA序列控制,但由DNA甲基化、组蛋白修饰和小RNA等调控,被称之为“软遗传”,对动、植物应对内、外因素(包括杂交、基因组互作、外界环境刺激等)具有很高的可塑性。上世纪50年代在玉米和高粱中报道的副突变现象就是一个经典的杂交诱导DNA甲基化变异跨代遗传的实例。副突变现象在动物小鼠中也有报道,但对于其发生的机制并未进行系统性的全基因组水平上的研究。
本研究在玉米自交系B73和Mo17的杂交种F1中鉴定到数千个受杂交诱导的DNA甲基化变异位点,其中有数百个位点的DNA甲基化状态可以一直传递到回交系第9代(回交6次及自交3次)(图1),被称为杂交诱导可跨代遗传的DNA甲基化。这些可传代DNA甲基化位点(表观位点,epiallele)的甲基化状态来自于其非轮回亲本,虽然DNA序列基本上(>99%)被替换为轮回亲本。并且主要发生在基因的上、下游附近,可能调控附近基因(表观基因,epigene)的表达。
图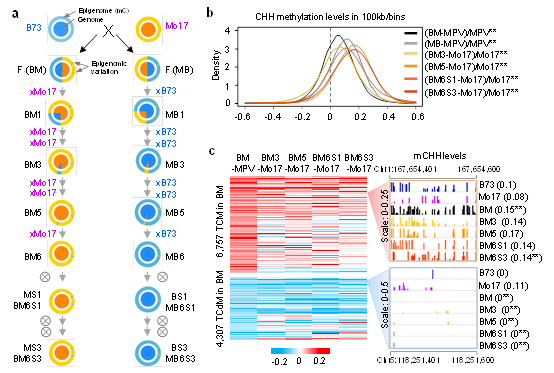
1:玉米杂交诱导的DNA甲基化变异在回交后代中多代遗传
进一步研究表明,这些表观位点与24-nt干扰小RNA(siRNAs)位点有关(图2)。线虫在外界食物刺激下,神经系统会产生一类可以连续传递至少4代的小RNA,并指导后代的弊害行为。在植物中,这些siRNA会通过RdDM途径诱导DNA非对称的胞嘧啶(CHH)甲基化,可以转变为稳定的甲基化并遗传。在玉米中,控制小RNA形成的副突变调控基因(MOP1)调节b1位点的副突变现象。本研究发现,在24-nt siRNA表达缺陷突变体的杂交种F1:mop1-1中,杂交诱导的DNA甲基化位点的甲基化变异在F1:mop1-1完全消失,表明24nt siRNA对这些位点的DNA甲基化变异建立和跨代遗传起到重要作用。因此,杂交诱导小RNA产生反式变化(trans-acting)的DNA甲基化位点,可以跨代遗传。该表观位点跨代遗传规律在野生玉米(大刍草)和玉米(W22)的种间杂交及回交组合中同样得到证明,也与24nt siRNA的表达位点(RdDM靶位点)有关。转录组表达研究表明,这些表观位点和表观基因的表达有关,很多表观基因与环境胁迫相关。一些副突变基因如b1控制色素形成也与环境胁迫相关。
综上所述,该研究通过系统的杂交、回交和多组学联合分析,揭示了玉米表观变异位点的形成和跨传代遗传的分子机制,以及表观基因表达对植物生长和环境响应的影响,为作物与动物育种提供新观点。
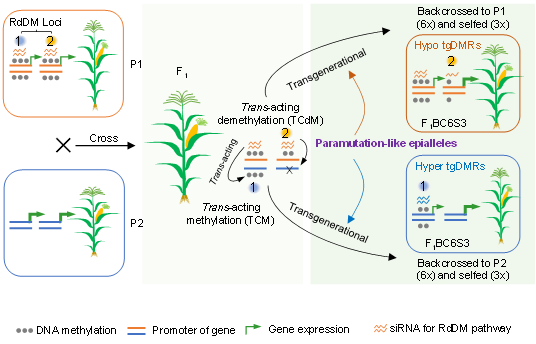
|
图2:玉米杂交诱导可传代DNA甲基化
beat365体育官方网站博士研究生曹帅为文章的第一作者,德克萨斯大学奥斯汀分校教授Z. Jeffrey Chen和beat365体育官方网站宋庆鑫教授为共同通讯作者,beat365平台王龙飞、韩同文、叶文雪、刘阳,和山西农业大学生科院生物中心孙毅研究员、伊利诺伊大学作物科学研究所Stephen P. Moose教授为共同作者。
文章链接: https://genomebiology.biomedcentral.com/articles/10.1186/s13059-022-02614-0
- 上一篇: 《Plant Biotechnology Journal》、《Journal of Experimental Botany》分别发表张红生课题组“OsHIPL1, a hedgehog-interacting protein-like 1 protein, increases seed vigor in rice”和“Identification of OsPK5 involved in rice glycolytic metabolism and GA/ABA balance for improving
- 下一篇: 《Genome Biology》发表宋庆鑫课题组“Open chromatin interaction maps reveal functional regulatory elements and chromatin architecture variations during wheat evolution”
